Équipe
VERA PANCALDI
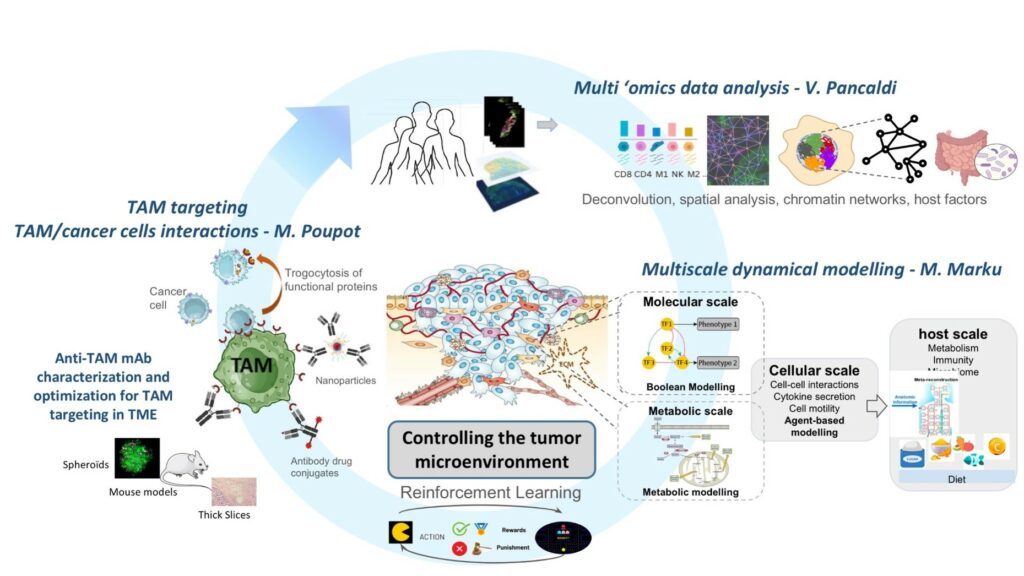
NetB(IO)² :
Network Biology for Immuno-oncology
Les spécificités
de notre axe de recherche
Notre laboratoire étudie la relation entre les patients et le cancer, en se focalisant sur la façon dont le système immunitaire et la tumeur interagissent. Nous cherchons à améliorer les thérapies qui exploitent cette interaction. Actuellement, ces traitements ne fonctionnent que sur certains patients et, souvent, pendant une période limitée avant qu’ils ne développent une résistance à la thérapie.
Notre objectif est de comprendre comment la variabilité des patients, des tumeurs et de leurs interactions affecte l’efficacité de ces thérapies. Nous utilisons des approches informatiques, notamment la science des données, la modélisation mathématique et surtout la théorie des réseaux, ainsi que plusieurs systèmes de modèles biologiques.
Les réseaux sont un cadre utile pour représenter des systèmes dans lesquels des relations telles que les interactions ou la similitude entre objets sont importantes. Tous les réseaux partagent certaines propriétés communes et il existe une vaste littérature sur la façon de les mesurer. La beauté est que notre compréhension d’un type de réseau peut nous aider à mieux comprendre un tout autre.
Nous appliquons des modèles de réseau pour étudier des systèmes aussi divers que les réseaux de similitude patient-patient, les réseaux d’interaction 3D de gènes dans le noyau et les réseaux d’interactions entre différents types de cellules dans une tumeur.
Il existe de nombreux contextes dans l’organisme qui impliquent un mélange hétérogène de types cellulaires. Dans les tissus, des relations complexes entre les types cellulaires établissent des «réseaux écologiques » dont les propriétés reflètent les équilibres qui définissent la santé par rapport à la maladie. Dans les tumeurs, les cellules cancéreuses sont entourées de populations cellulaires extrêmement hétérogènes, notamment des cellules stromales et immunitaires, dont les schémas spatiaux et les interactions spécifiques ont un fort impact sur la réponse aux thérapies et l’issue de la maladie pour les patients.
La simulation des interactions complexes de ces cellules dans nos ordinateurs, tant au niveau moléculaire que intercellulaire, offre la possibilité d’étudier ces systèmes dans une multitude de scénarios. C’est un excellent moyen de formuler de nouvelles hypothèses et de mieux utiliser nos ressources expérimentales.
Nos principaux objectifs de recherche sont les suivants
- Caractériser les populations cellulaires et leurs interactions dans le microenvironnement tumoral (MET) en prenant en compte les réseaux de communication intercellulaire et les modèles spatiaux extraits de la transcriptomique (à la fois en masse et à cellule unique) ou de l’omique spatiale et de l’imagerie produites à partir d’échantillons de patients ainsi que de leur données épigénomiques.
- Simuler les processus cellulaires dynamiques et les interactions intercellulaires dans le MET impliquant des cellules cancéreuses, immunitaires et stromales, en exploitant des modèles mathématiques à différentes échelles (moléculaire, cellulaire, etc.) de modèles de cancer in vitro (2D/3D) et in vivo.
- Développer des stratégies pour contrôler le microenvironnement tumoral afin d’améliorer la sensibilité des cellules cancéreuses aux traitements en exploitant un anticorps monoclonal breveté en interne ciblant spécifiquement les macrophages associés aux tumeurs, en identifiant de nouvelles stratégies de reprogrammation du MET et en élargissant notre vision aux caractéristiques des patients (aptitude immunitaire, microbiome, expositions, etc.)
Biologie computationnelle
Théorie des Réseaux
Hétérogénéité et Microenvironnement tumoral
Système Immunitaire
Epigénomique
Transcriptomique spatiale et à cellule unique
Architecture du génome
Modèles Mathématiques
DES PROJETS
DE RECHERCHE
LES FOCUS
DE L’ÉQUIPE
Découvrir
Comprendre
En live
Just a few hours to go for early bird registration for our @netbiomed2025 symposium, join us in Maastricht on June 2nd. Check out our website for registration instructions #AI #DigitalTwins @net_science @NetSciConf
3 days before @NetSci2025 early bird ends: Don't miss your chance to attend @netbiomed2025. Please check out our website for details 7 Amazing speakers https://netbiomed2025.github.io AI Digital twins @BSC_CNS @CNIOStopCancer @esantapau @CamNetNet @net_science @ConfCompSys @SysBioCurie
PRODUCTIONS SCIENTIFIQUES
PUBLICATIONS 2025
PUBLICATIONS 2024
PUBLICATIONS 2023
PUBLICATIONS 2022
PUBLICATIONS 2021
PUBLICATIONS 2020
PUBLICATIONS 2019
LES MEMBRES DE L’ÉQUIPE
LES PARTENAIRES & FINANCIERS

Centre de Recherches en Cancérologie de Toulouse (Oncopole)
Toulouse – FR
CRCT-Oncopole Suivre 1,433 1,279
Compte officiel du Centre de Recherches en Cancérologie de Toulouse
🇬🇧Cytidine deaminase-dependent mitochondrial biogenesis as a potential vulnerability in pancreatic cancer cells. ImPact team @CordelierPierre article from @crctoncopole published in @CommsBio
➡️https://www.crct-inserm.fr/en/targeting-the-energy-productio...
Nous contacter
05 82 74 15 75
Envie de rejoindre
L’équipe du CRCT ?