Équipe
STEFANIA MILLEVOI
RNAreg :
Protéines De Liaison À L’ARN Et Stress Génotoxique
Les spécificités
de notre axe de recherche
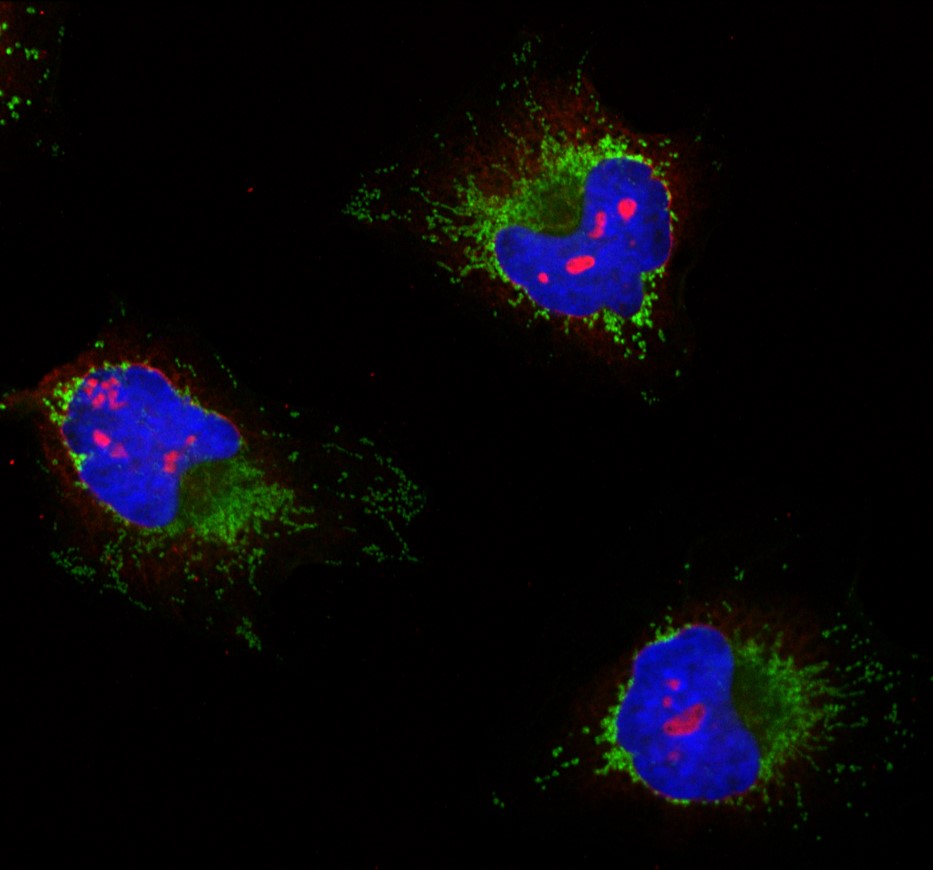
Notre équipe s’intéresse à la régulation de l’expression des gènes du cancer au niveau post-transcriptionnel.
En particulier, nous étudions les gènes impliqués dans les processus cellulaires clés de la réponse au stress génotoxique associés à l’émergence de mécanismes de résistance aux thérapies anticancéreuses telles que la chimio/radiothérapie, mais aussi l’immunothérapie. Le développement de ce projet d’envergure est assuré par la mutualisation d’expertises distinctes mais complémentaires dans les domaines des ARN codants (Anne Cammas, Hervé Prats, Stefania Millevoi) et non-codants (Estelle Espinos), et dans les processus cellulaires de réponse aux dommages à l’ADN, notamment l’autophagie, l’apoptose et le cycle cellulaire (Stéphane Manenti et Estelle Espinos).
Bien que nos projets découlent de questions fondamentales sur la régulation de l’ARN et la réponse au stress génotoxique, en particulier l’autophagie, notre ambition ultime est de fournir des informations sur leur pertinence clinique dans les cancers de la tête et du cou (HNSCC), une maladie fréquente et agressive, associée à un taux élevé de récidive et de mortalité.
Pour cela, notre équipe bénéficie de l’expertise de Sébastien Vergez, oto-rhino-laryngologiste et chirurgien cervico-facial, ainsi que de la synergie avec le comité ORL et le bureau d’études cliniques de l’IUCT-O (Thomas Filleron).
Le programme de recherche intègre des analyses bioinformatiques et des approches expérimentales, qui combinent des analyses transcriptomiques/protéomiques à grande échelle avec des études plus mécanistiques sur des gènes candidats, ainsi que des analyses réalisées sur les échantillons primaires prélevés chez les patients.
Les connaissances acquises dans le cadre de ce projet, reliant la molécule de l’ARN au patient, devraient permettre de mettre en lumière de nouveaux concepts pour le développement de stratégies thérapeutiques basées sur l’ARN ciblant l’autophagie dans les cancers HNSCC.
Cancers de la tête et du cou (HNSCC)
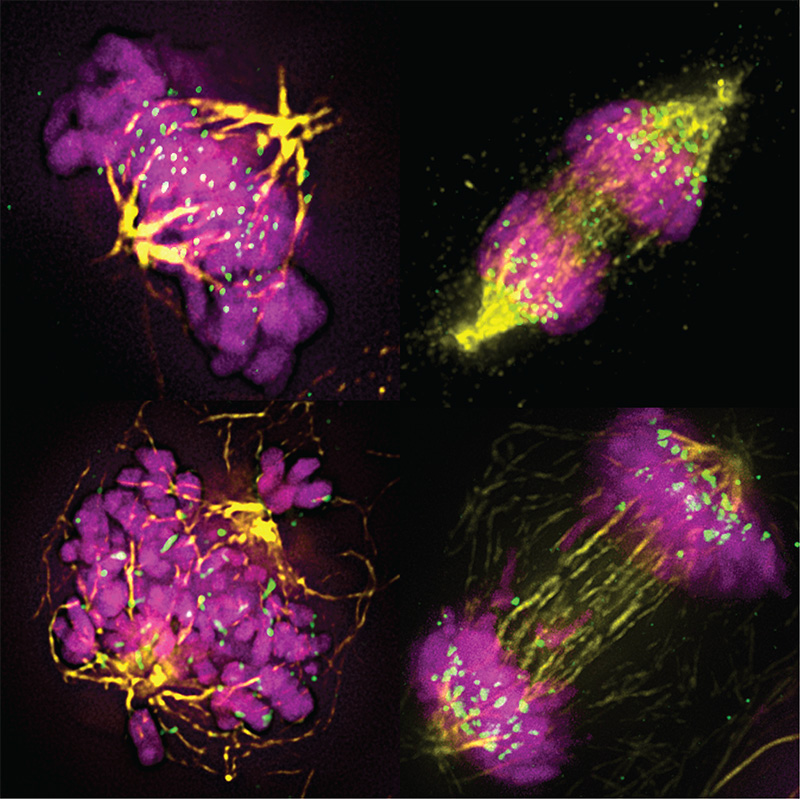
Réponse aux dommages à l’ADN
Mécanismes moléculaires de la résistance thérapeutique
Autophagie
Régulations post-transcriptionnelles
Epitranscriptomique
Traduction des ARNm
Protéines de liaison à l’ARN
Structures d’ARN de type G-quadruplexe
Approches « omiques »
Bioinformatique
DES PROJETS
DE RECHERCHE
Structures ARN G-quadruplexes & adaptation cellulaire au stress
Anne Cammas
Régulateurs de la réponse aux dommages à l’ADN dans le contrôle de la post-transcription
Stéphane Manenti
Lien entre ARN & autophagie
Estelle Espinos
Protéines de liaison à l’ARN, autophagie & réponse aux thérapies dans les HNSCC
Sébastien Vergez
LES FOCUS
DE L’ÉQUIPE
Découvrir
Comprendre
Participer
Aucun résultat
La page demandée est introuvable. Essayez d'affiner votre recherche ou utilisez le panneau de navigation ci-dessus pour localiser l'article.
En live
PRODUCTIONS SCIENTIFIQUES
PUBLICATIONS 2024
PUBLICATIONS 2023
PUBLICATIONS 2022
PUBLICATIONS 2021
PUBLICATIONS 2020
PUBLICATIONS 2019
LES MEMBRES DE L’ÉQUIPE
LES PARTENAIRES & FINANCIERS

Centre de Recherches en Cancérologie de Toulouse (Oncopole)
Toulouse – FR
CRCT-Oncopole Suivre 1,220 1,117
Compte officiel du Centre de Recherches en Cancérologie de Toulouse
Castelnau-Montratier célèbre la lutte contre le cancer. Jeudi 24 août 2023 conférence avec Frédéric Chibon responsable de l'équipe @OncoSarc du @crctoncopole accompagné de Gwenaël Ferron et Martin Gauthier oncologue @IUCTOncopole
Découvrir l'équipe
➡️https://www.crct-inserm.fr/oncosarc/
Nous contacter
05 82 74 15 75
Envie de rejoindre
L’équipe du CRCT ?