La transcription dans l’oncogenèse et la réponse aux traitements anticancéreux
Agnese Cristini
Notre domaine de recherche
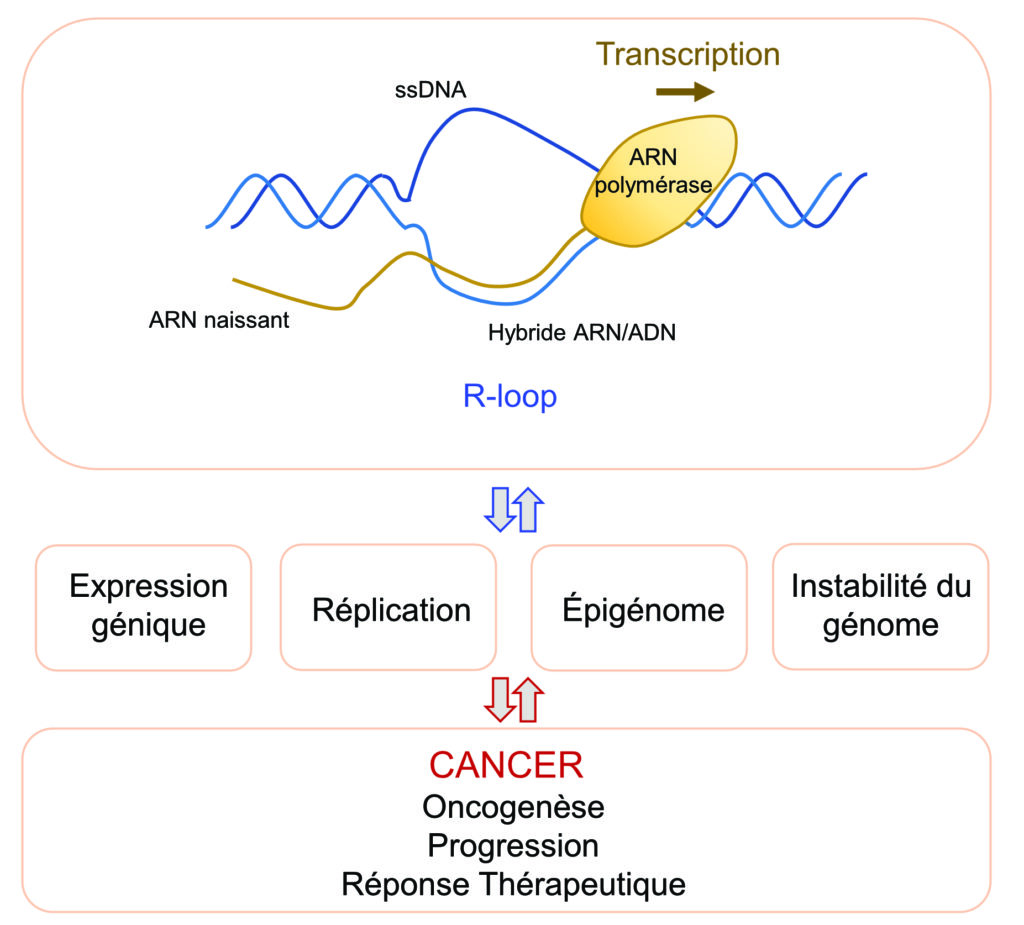
Les altérations génétiques et épigénétiques entraînent la dérégulation de l’expression génique, qui est une caractéristique des cancers. Les cellules cancéreuses sont « dépendantes » de ces programmes transcriptionnels dérégulés qui participent au maintien de la croissance et de la survie de la tumeur. La transcription est aussi associée à la formation de structures alternatives d’acide nucléique, appelées R-loops, qui sont constituées d’un hybride ARN/ADN et d’un ADN simple brin. Ces structures régulent et sont régulées par des processus cellulaires fondamentaux, dont l’expression des gènes, la réplication et le maintien de la stabilité génomique et épigénomique, qui sont tous impliqués dans le cancer.
Nos recherches visent à étudier les mécanismes moléculaires par lesquels la transcription participe à la survenue du cancer, à son évolution et à la réponse aux thérapies anticancéreuses. Pour cela, nous utilisons une combinaison d’approches génomiques, biochimiques, cellulaires et moléculaires.
Si vous êtes intéressés à nous rejoindre, contactez-nous par mail (agnese.cristini@inserm.fr) pour plus d’informations.
Mots clés:
- Cancer
- Transcription
- R-loop
- Expression génique
- Instabilité du génome
- Dommages à l’ADN
- Oncogenèse
- Thérapie anticancéreuse
- Thérapie ciblée
- Chromatine
- Épigénome
Membres du groupe
- Eglantine Faget, doctorante
- Emma Lehir, doctorante
- Anaïs Pouget, étudiante en Master 1
Alumni
- Maïna Vienne, ingénieure d’étude en bioinformatique
- Elisa Spielmann, étudiante en Master 2
- Lara Keszleri, étudiante Erasmus
- Elisona Shyti, étudiante Erasmus
- Ana Alvarez, doctorante invitée
- Stella Kristensen, étudiante Erasmus
- Jacqueline Mohr, étudiante DAAD
- Fanny Wandersleb, étudiante DAAD
- Jakob Freudenberger, étudiant DAAD
Autres membres de l’équipe impliqués
- Olivier Sordet, PhD, HDR, Chercheur (CRCN)
- Olivier Calvayrac, PhD, Chercheur (CRCN)
- Mathéa Géraud, Assistante Hospitalo-Universitaire
- Andrea Carla Ajello, doctorante
Publications sélectionnées
STAR Protoc. 2025 Mar 21;6(1):103662. doi: 10.1016/j.xpro.2025.103662. PMID: 40023839.
Geraud M, Fernandez Martinez L, Ajello AC, Cristini A#, Sordet O#.
Protocol for single-cell analysis of DNA double-strand break production and repair in cell-cycle phases by automated high-content microscopy. *
#Co-corresponding authors.
Nat Genet. 2023 Oct;55(10):1721-1734. doi: 10.1038/s41588-023-01504-w. PMID: 37735199
McCann JL*, Cristini A*, Law EK, Lee SY, Tellier M, Carpenter MA, Beghè C, Kim JJ, Sanchez A, Jarvis MC, Stefanovska B, Temiz NA, Bergstrom EN, Salamango DJ, Brown MR, Murphy S, Alexandrov LB, Miller KM, Gromak N, Harris RS.
APOBEC3B regulates R-loops and promotes transcription-associated mutagenesis in cancer.
*Co-first authors.
Nat Commun. 2022 May 26;13(1):2961. doi: 10.1038/s41467-022-30604-0. PMID: 35618715
Cristini A, Tellier M, Constantinescu F, Accalai C, Albulescu LO, Heiringhoff R, Bery N, Sordet O, Murphy S, and Gromak N.
RNase H2, mutated in Aicardi-Goutieres syndrome, resolves co-transcriptional R-loops to prevent DNA breaks and inflammation.
Cell Rep. 2019 Sep 17;28(12):3167-3181.e6. doi: 10.1016/j.celrep.2019.08.041. PMID: 31533039
Cristini A, Ricci G, Britton S, Salimbeni S, Huang SN, Marinello J, Calsou P, Pommier Y, Favre G, Capranico G, Gromak N, Sordet O.
Dual Processing of R-Loops and Topoisomerase I Induces Transcription-Dependent DNA Double-Strand Breaks.
Cell Rep. 2018 May 8;23(6):1891-1905. doi: 10.1016/j.celrep.2018.04.025. PMID: 29742442
Cristini A*, Groh M*, Kristiansen MS, Gromak N.
RNA/DNA Hybrid Interactome Identifies DXH9 as a Molecular Player in Transcriptional Termination and R-Loop-Associated DNA Damage.
*Co-first authors.