
ALEXIS COULLOMB
INVESTIGATEURS ASSOCIES : JULIE BORDENAVE & NINA VERSTRAETE
Etude De L’organisation Spatiale Du Microenvironnement Tumoral Et Approches Multi-Omiques
Les tumeurs solides sont constituées de différents types cellulaires, telles que les cellules tumorales ou les cellules immunitaires pro- ou anti-tumorales, dont les proportions et l’organisation spatiale jouent un rôle décisif sur la progression des tumeurs ou leur réponse aux traitements. Par ailleurs, de nombreuses méthodes ont été développées pour produire des cartes d’abondance de protéines ou d’ARNm de cellules uniques, telles que l’immunofluorescence multiplexée ou l’ARN-FISH cyclique.
Notre équipe développe des outils pour analyser les données hautement complexes générées par ces méthodes, et leur représentation en réseaux est une approche efficace pour analyser ces données omiques spatiales. La librairie tysserand permet de reconstruire les réseaux spatiaux à partir des expériences de omiques spatiallement résolues, et la librairie mosna (Multi Omics Spatial Networks Analysis) fournit des méthodes pour analyser ces réseaux spatiaux pour étudier les interactions entre cellules et découvrir des communautés de cellules potentiellement intéressantes cliniquement.
De plus, nous implémentons des protocoles FISH pour produire des données omiques spatiales concernant l’immuno-oncologie.
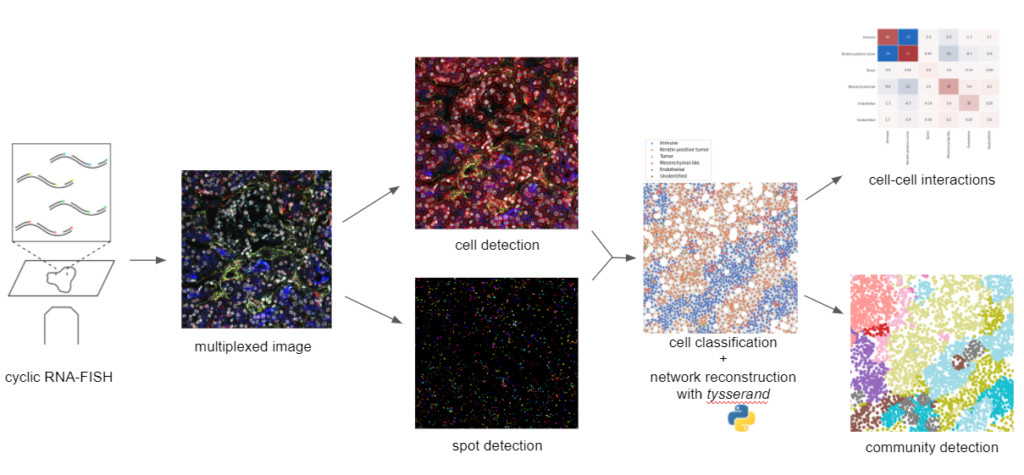
Pipeline de transcriptomique spatial à cellule unique : la FISH cyclique d’ARN consiste en des cycles successifs d’hybridation de sondes conjuguées à un fluorophore à des ARN d’intérêt, suivis d’une déshybridation pour les cycles suivants. Les données résultantes sont une image multiplex, avec autant de canaux de fluorescence que d’espèces d’ARN ciblées par les sondes FISH. Après la détection des cellules individuelles par segmentation cellulaire et des molécules d’ARN par détection de points, ces informations sont combinées pour définir les phénotypes des cellules individuelles, et pour reconstruire le réseau tissulaire spatial avec la bibliothèque tysserand. Ce réseau spatial est ensuite utilisé pour inspecter les interactions cellule-cellule et trouver des voisinages spatiaux (ou “niches”) d’intérêt clinique potentiel.
Financements : Janssen
Chaire d’ informatique en oncologie du CRCT (Fondation Toulouse Cancer Sante, Inserm et Pierre Fabre)
Pancaldi and Coullomb 2021
https://academic.oup.com/bioinformatics/advance-article-abstract/doi/10.1093/bioinformatics/btab490/6313163?redirectedFrom=fulltext
Github : https://github.com/VeraPancaldiLab/tysserand.

