
Leila Khajavi
INVESTIGATEURS ASSOCIES : JACOBO SOLORZANO, ALEXIS HUCTEAU, MIGUEL MADRID-MENCIA, ALEXIS COULLOMB & NINA VERSTRAETE
Déconvolution : (Description De La Composition Du Microenvironnement Tumoral) Vers La Médecine Personnalisée
L’accroissement des connaissances sur la classification des différents cancers et leurs traitements permettent de se diriger vers une médecine personnalisée, où chaque patient pourra recevoir un traitement différent qui lui correspond vraiment. Notre équipe se focalise sur la découverte de nouvelles cibles liées à l’environnement tumoral qui puissent être exploitées à des fins thérapeutiques. Nous explorons aussi la création d’algorithmes d’intelligence artificielle permettant de prédire la réponse d’un patient à un traitement donné, en fonction de ses antécédents médicaux.
C’est pourquoi notre équipe collabore avec de nombreux partenaires cliniques et industriels dans le but de découvrir de nouvelles approches thérapeutiques pour les patients atteints de cancer du poumon.
Dans ce but, la détermination de la composition cellulaire d’un échantillon est une priorité pour la recherche en oncologie. En s’appuyant sur l’importante quantité de données disponible dans le domaine scientifique public, notre équipe fait preuve d’approches innovantes regroupant ces données omiques afin de générer de nouvelles et meilleures signatures pour les algorithmes de déconvolution supervisée.
Il s’agit d’une technique informatique permettant de déterminer la composition cellulaire d’un échantillon (tumoral), afin de prédire la réponse aux traitements d’immunothérapie par l’utilisation de modèles d’IA.
Notre principale collaboration dans ce domaine s’articule autour du projet LungPredict (LungPredict: improve and personalize immuno-therapies – CRCT). Grâce, entre autres, aux analyses de déconvolution, le but est d’extraire et d’exploiter l’information génétique présente dans les échantillons tumoraux de patients atteints de cancer du poumon. Les données que nous en tirons nous permettent de rechercher des protéines et gènes qui peuvent être de potentielles cibles thérapeutiques pour les approches de médecine personnalisée.
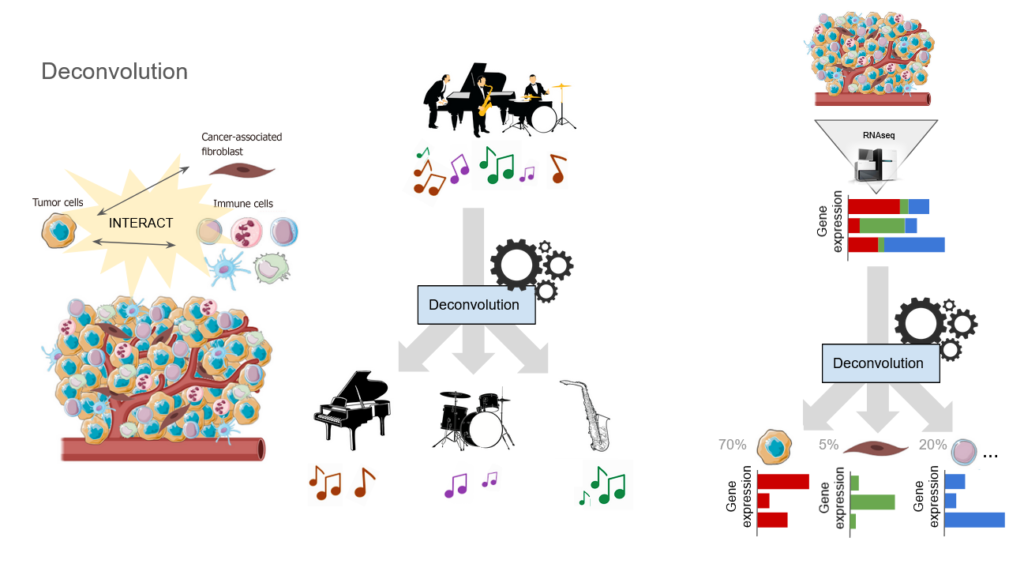
La déconvolution est une méthode informatique permettant d’identifier la composition d’échantillons mixtes à partir de mesures globales (telles que l’expression génique ou la méthylation de l’ADN) sur la base des profils connus de types cellulaires spécifiques. Nous l’utilisons pour identifier les différents types de cellules présents dans les échantillons de tumeurs.
Github : https://github.com/VeraPancaldiLab/GEMDeCan
Chaire d’ informatique en oncologie du CRCT (Fondation Toulouse Cancer Sante, Inserm et Pierre Fabre)

