Équipe
Frédéric Chibon
ONCOSARC :
Oncogenèse des sarcomes
Les spécificités
de notre axe de recherches
Sarcome
Différenciation
Diagnostic
Génome
Oncogenèse
Pronostic
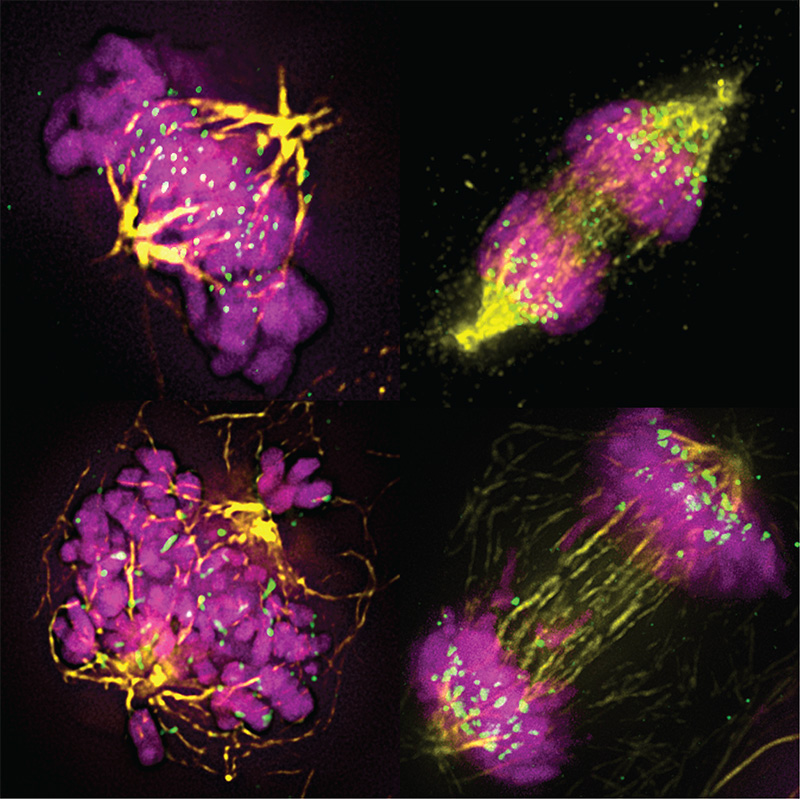
Chromosome
Metastase
Cellule tumorale hybride
Mitose
Traitement
Hétérogénéité
DES PROJETS
DE RECHERCHE
Oncogenèse des sarcomes
Frédéric Chibon
LES FOCUS
DE L’ÉQUIPE
Découvrir

Comprendre
Participer
Aucun résultat
La page demandée est introuvable. Essayez d'affiner votre recherche ou utilisez le panneau de navigation ci-dessus pour localiser l'article.
En live
Nous recrutons!
Le groupe "Oncogenèse des sarcomes" de l'ONCOPOLE de Toulouse cherche à se renforcer, pour développer de nouveaux axes. Recherchons CR/DR INSERM.
Faites passer l'info, contactez moi!
Détail sur le site Rh de l'INSERM:https://rh.inserm.fr/Pages/default.aspx
PRODUCTIONS SCIENTIFIQUES
PUBLICATIONS 2024
PUBLICATIONS 2023
PUBLICATIONS 2022
PUBLICATIONS 2021
PUBLICATIONS 2020
PUBLICATIONS 2019
LES MEMBRES DE L’ÉQUIPE
LES PARTENAIRES & FINANCIERS

Centre de Recherches en Cancérologie de Toulouse (Oncopole)
Toulouse – FR
CRCT-Oncopole Suivre 1,220 1,117
Compte officiel du Centre de Recherches en Cancérologie de Toulouse
Castelnau-Montratier célèbre la lutte contre le cancer. Jeudi 24 août 2023 conférence avec Frédéric Chibon responsable de l'équipe @OncoSarc du @crctoncopole accompagné de Gwenaël Ferron et Martin Gauthier oncologue @IUCTOncopole
Découvrir l'équipe
➡️https://www.crct-inserm.fr/oncosarc/
Nous contacter
05 82 74 15 75
Envie de rejoindre
L’équipe du CRCT ?