Kartezio : une approche entièrement transparente de l’analyse d’images biomédicales basée sur l’IA
Intelligence artificielle,
traitement d’images,
apprentissage automatique,
, immuno-oncologie,
cellules T cytotoxiques.
Brienne McKenzie et Salvatore Valitutti équipe DynAct Dynamique moléculaire des interactions lymphocytaires
En médecine numérique, les algorithmes d’intelligence artificielle (IA) utilisés pour l’analyse d’images médicales doivent non seulement être très précis, mais aussi totalement transparents et interprétables, afin d’instaurer la confiance, de garantir le respect des normes éthiques et de faciliter la surveillance réglementaire. De nombreux algorithmes performants basés sur l’apprentissage profond sont néanmoins intrinsèquement difficiles à interpréter en raison de leur nature de “boîte noire”, ainsi que de leur coût de calcul et de la nécessité de disposer d’énormes ensembles de données pour l’entraînement. Dans cette étude, nous présentons Kartezio, une approche d’IA évolutionnaire, la première de sa catégorie, qui génère des algorithmes d’analyse d’images “boîte blanche” entièrement transparents et facilement interprétables. Kartezio a égalé les performances des approches de Deep Learning les plus récentes sur des tâches de traitement d’images spécifiques, tout en ne nécessitant qu’une poignée d’images pour s’entraîner. Nous avons validé cette approche révolutionnaire en utilisant un panel d’études de cas en immuno-oncologie, allant de la délimitation de tumeurs entières en pathologie à la détection de particules subcellulaires en utilisant la microscopie à super-résolution. Nous avons démontré que Kartezio est précis, robuste, flexible, rapide et totalement transparent pour l’homme, ce qui change la donne dans l’analyse d’images biomédicales basée sur l’IA.
Ce travail fournit une nouvelle approche entièrement transparente basée sur l’IA pour l’analyse d’images médicales, avec de vastes applications potentielles dans toutes les disciplines cliniques, y compris la pathologie et la radiologie, ainsi que la microscopie. Nous avons validé notre approche unique dans de multiples études de cas en immuno-oncologie, y compris la délimitation de tumeurs entières et la détection de particules subcellulaires cytotoxiques. Ces études de cas soulignent l’impact de cette approche d’IA très précise et totalement transparente dans le contexte clinique et la diversité des applications pour lesquelles elle est pertinente.
Les approches décrites peuvent potentiellement être appliquées à l’analyse d’images biomédicales de différentes sources et résolutions. Elles peuvent être utilisées dans la recherche, les études cliniques et le diagnostic.
La demande de brevet suivante a été déposée par KC, SV, SCB, DW, HL et BM : “K. Cortacero et al. filed patent ; EP 22307041.8”.
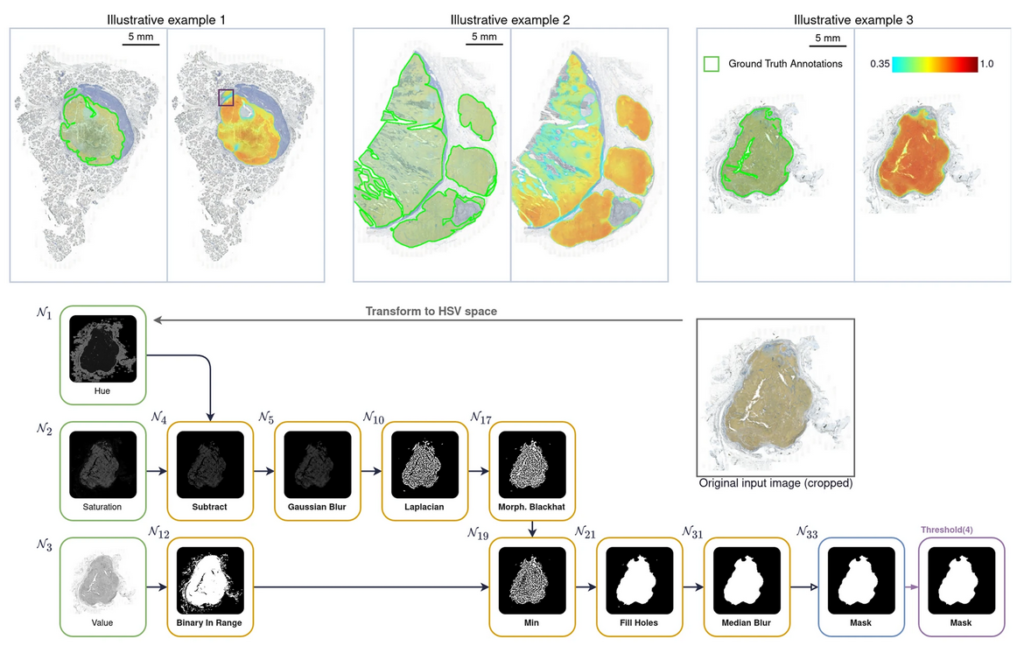
EN HAUT : trois exemples de nodules de mélanome annotés manuellement par un expert humain (lignes vertes) ou automatiquement à l’aide de pipelines générés par Kartezio (carte thermique multicolore).
EN BAS : un exemple de pipeline de segmentation d’image entièrement transparent, étape par étape, généré par Kartezio.
Découvrir l’article publié
Nat Commun. 2023 Nov 6;14(1):7112.doi: 10.1038/s41467-023-42664-x.
Evolutionary design of explainable algorithms for biomedical image segmentation
Kévin Cortacero, Brienne McKenzie, Sabina Müller, Roxana Khazen, Fanny Lafouresse, Gaëlle Corsaut, Nathalie Van Acker, François-Xavier Frenois, Laurence Lamant, Nicolas Meyer, Béatrice Vergier, Dennis G Wilson, Hervé Luga, Oskar Staufer, Michael L Dustin, Salvatore Valitutti, Sylvain Cussat-Blanc
Collaborations et partenariats
Cette recherche est le fruit d’une collaboration interdisciplinaire entre des immunologistes travaillant au Centre de recherche sur le cancer de Toulouse (CRCT) et au Kennedy Institute de l’Université d’Oxford (membres du consortium ATTACK financé par le Conseil européen de la recherche), des informaticiens travaillant à l’Institut de recherche en informatique de Toulouse (IRIT) et des dermatopathologistes et dermatoncologues travaillant à l’Institut universitaire du cancer de Toulouse. Le travail a également été réalisé en utilisant les ressources HPC du centre de supercalcul CALMIP de Toulouse.

Centre de Recherches contre le Cancer de Toulouse (Oncopole)
Toulouse - FR
Nous contacter
+33 5 82 74 15 75
Nous rejoindre ?




